我有以下代码对数据执行分层聚类:
Z = linkage(data,method='weighted')
plt.subplot(2,1,1)
dendro = dendrogram(Z)
leaves = dendro['leaves']
print leaves
plt.show()
无论如何,在登革热所有的集群都有相同的颜色(蓝色)。有没有一种方法来使用不同的颜色相对于集群之间的相似性?
查看文档,看起来您可以传递link\u color\u func关键字或color\u threshold关键字以获得不同的颜色。
编辑:
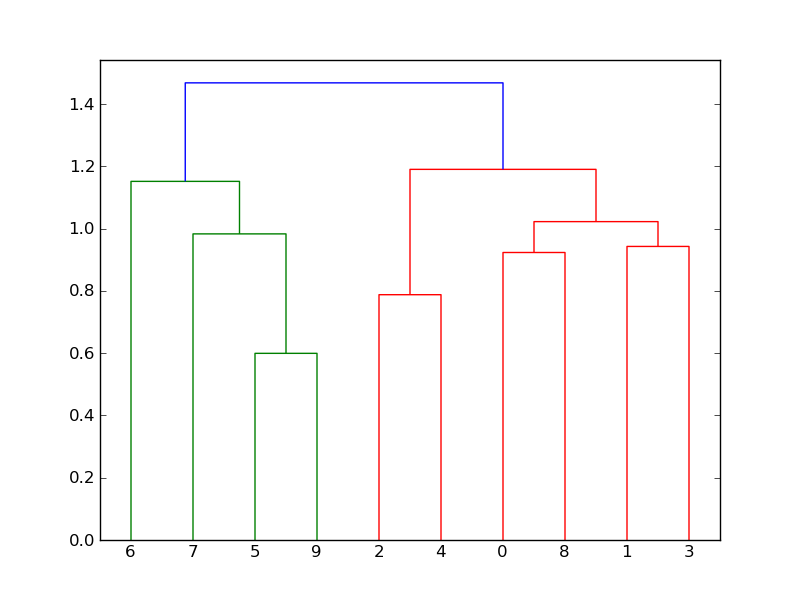
树状图着色方案的默认行为是,给定color_threshold=0.7*max(Z[:,2]),将集群节点k下面的所有子代链接着色为相同的颜色,如果k是第一个节点低于切割阈值;否则,连接距离大于或等于阈值的节点的所有链接都被染成蓝色[来自文档]。
这到底是什么意思?如果你看树状图,不同的星系团连接在一起。两个集群之间的“距离”是它们之间链接的高度。color_threshold是新集群的高度,低于该高度将是不同的颜色。如果你所有的集群都是蓝色的,那么你需要提高你的color_threshold。例如,
In [48]: mat = np.random.rand(10, 10)
In [49]: z = linkage(mat, method="weighted")
In [52]: d = dendrogram(z)
In [53]: d['color_list']
Out[53]: ['g', 'g', 'b', 'r', 'c', 'c', 'c', 'b', 'b']
In [54]: plt.show()
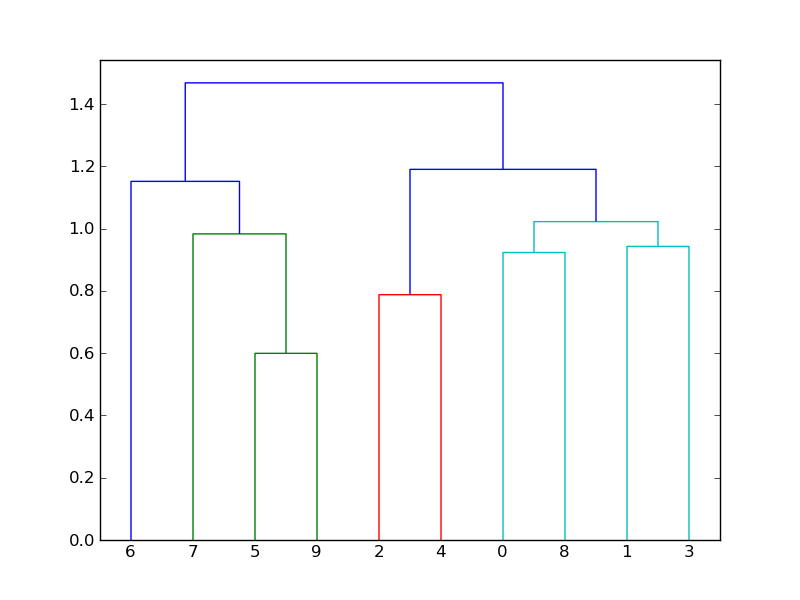
我可以通过以下方式检查默认的颜色\u阈值
In [56]: 0.7*np.max(z[:,2])
Out[56]: 1.0278719020096947
如果我降低color_threshold,我会得到更多的蓝色,因为更多的链接的距离大于新的color_threshold。你可以直观地看到这一点,因为0.9以上的所有链接现在都是蓝色的:
In [64]: d = dendrogram(z, color_threshold=.9)
In [65]: d['color_list']
Out[65]: ['g', 'b', 'b', 'r', 'b', 'b', 'b', 'b', 'b']
In [66]: plt.show()
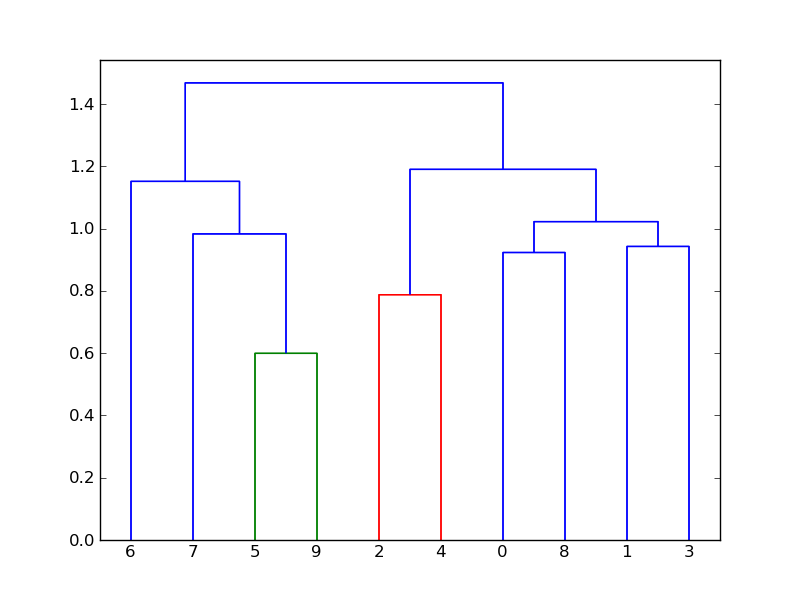
如果我将color\u阈值增加到1.2,则1.2下面的链接将不再为蓝色。此外,青色和红色链接将合并为一种颜色,因为它们的父链接位于1.2下面: